Geneious Prime是專業(yè)的分子生物學和序列分析工具,從事相關行業(yè)的用戶就會需要用到它,本次給大家?guī)淼氖亲钚碌?021破解版,激活后就可以免費使用。下面還有這詳細的安裝破解教程。軟件提供了全面的生物信息學分析工具,支持序列比對、序列觀看、PCR引物設計等多種功能,還可以模擬各個步驟的分子克隆操作。不管是生物制藥、生命科學還是學術研究都會用到它。
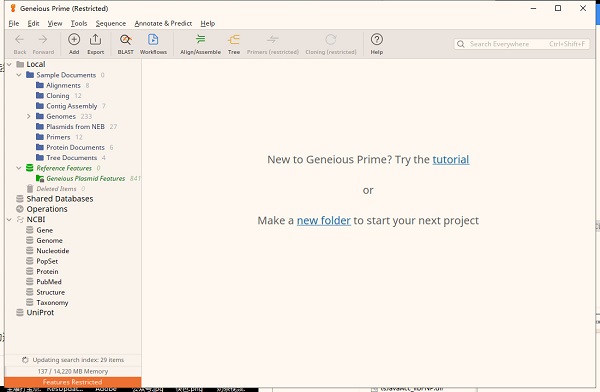
安裝方法
1、首先從本站下載數(shù)據(jù)包然后解壓,得到安裝程序“Geneious_Prime_win64_with_jre.msi”,鼠標雙擊運行進入安裝向?qū)c擊“next”進入下一步
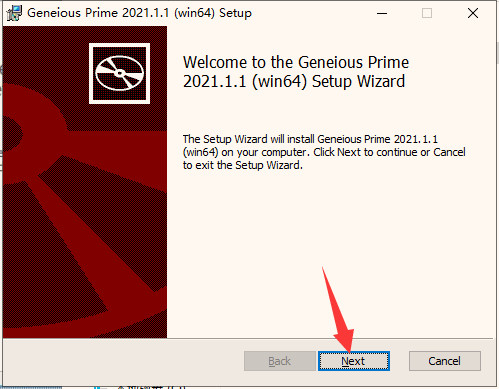
2、選擇第一項“I accept the terms of the license agreement”(我接受許可協(xié)議的條款),再點擊“next”進入下一步|

3、選擇安裝位置,默認路徑為“C:\Program Files\Geneious Prime\”,建議最好不要安裝到系統(tǒng)盤(C盤)

4、勾選“create a dessktop icon”(創(chuàng)建一個桌面圖標)

5、軟件安裝需要一些時間請耐心等待即可

6、當安裝完成后點擊“finish”即可退出安裝向?qū)?/p>

7、安裝完成后先不要運行,回到剛才下載的數(shù)據(jù)包中將破解補丁復制道軟件的安裝目錄中替換原文件即可
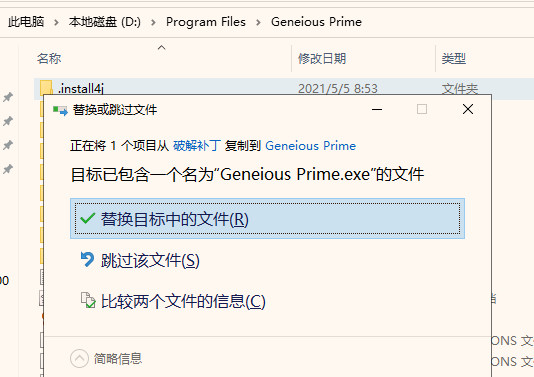
8、最后軟件軟件即可開始免費使用,破解完成

軟件特色
1、NGS預處理
導入Illumina,PacBio和NanoPore讀取
修剪,過濾和解復用單端和成對端數(shù)據(jù)
合并配對的讀
重復數(shù)據(jù)刪除
錯誤糾正和規(guī)范化
濾除嵌合體
學到更多
2、制圖和重新組裝
只需在業(yè)界領先的制圖算法和從頭組裝算法之間切換
支持匯編Sanger和NGS數(shù)據(jù),包括任何長度的Illumina,PacBio和Oxford納米孔讀取,包括雙端讀取和混合裝配
組裝微生物基因組,質(zhì)粒和其他環(huán)狀序列時產(chǎn)生環(huán)狀重疊群
基因組比較和MAUVE基因組比對結束
映射程序包括BBMap,Minimap2,Bowtie2和TopHat
從頭組裝算法,包括SPAdes,F(xiàn)lye,MIRA,Tadpole和Velvet
3、變體調(diào)用和表達分析
使用FreeBayes調(diào)用SNP /變體
使用同步的基因組視圖對表格結果進行實時過濾
在映射的RNA-seq數(shù)據(jù)上計算和比較表達水平
使用PCA和火山圖進行可視化

4、序列分析
修剪,組裝和查看Sanger測序跟蹤文件
更正基礎調(diào)用并創(chuàng)建共識序列
注釋主題,ORF和重復
預測基因和結構元素
通過針對數(shù)據(jù)庫的相似性搜索進行實時注釋
快速翻譯選擇內(nèi)容,或顯示注釋或所選框架的翻譯
動態(tài)圖和統(tǒng)計信息,用于序列特性,例如pI,分子量,熔點,AA組成等
5、序列比對
DNA或蛋白質(zhì)的多重和成對序列比對,包括全基因組比對
與包括Aligner,MUSCLE,MAFFT,Clustal Omega,MAUVE和LastZ在內(nèi)的可信算法保持一致
使用實時翻譯和突出顯示查看和編輯路線
6、系統(tǒng)發(fā)育學
使用Geneious Prime 2021樹構建器,MrBayes,PAUP *,PhyML,RAxML等構建樹
可視化,編輯和標記您的樹
交互式距離矩陣查看器
出版物質(zhì)量出口
7、微衛(wèi)星分析
導入原始ABI跟蹤文件
修剪,預測和手動調(diào)整峰
Bin進入等位基因
產(chǎn)生等位基因調(diào)用的表格輸出
8、分子克隆
查看質(zhì)粒圖譜,自動注釋載體以及帶有注釋的復制粘貼序列
一步式GoldenGate(IIS類型)和限制克隆
基于同源性的克隆,包括Gibson,GeneArt和In-Fusion
TOPO克隆
克隆操作的父母/后代血統(tǒng)追蹤
密碼子優(yōu)化和反向翻譯
沉默突變分析以發(fā)現(xiàn)潛在的限制性酶切位點
模擬PCR,消化和連接
CRISPR站點查找器
9、底漆設計
自動設計針對任何目標區(qū)域或整個序列的PCR和測序引物以及雜交探針
在序列視圖中輕松添加引物
設計基本和簡并PCR引物
在設計過程之前,之中或之后添加和刪除引物序列的延伸
引物特異性測試,以檢查模板序列上是否有其他結合位點
篩選物理性質(zhì),發(fā)夾和引物二聚體
以FASTA,電子表格或GenBank格式拖放引物
10、數(shù)據(jù)管理與協(xié)作
拖放導入文件和文件夾,包括 Vector NTI數(shù)據(jù)庫
將電子表格中的元數(shù)據(jù)導入序列和其他文檔
智能NGS導入–一步導入任何種類的SAM,BAM,GFF,BED和VCF文件
直觀的基于文件夾的項目組織
無縫集成共享數(shù)據(jù)庫
快速搜索數(shù)據(jù)庫中的所有序列和元數(shù)據(jù)
廣泛的出口選擇
11、搜索和爆炸
直接訪問NCBI公共BLAST數(shù)據(jù)庫
私人本地數(shù)據(jù)庫的自定義BLAST
集成搜索外部數(shù)據(jù)庫,包括GenBank和UniProt
將序列直接上傳到GenBank
在PubMed中搜索文獻
針對本地或共享數(shù)據(jù)庫的高級搜索
12、工作流程
使用可視化編輯器創(chuàng)建用于自動化批量分析的工作流
超過20種內(nèi)置工作流,用于執(zhí)行管道,包括將變體應用于參考序列,圖譜讀取然后查找SNP和隨機采樣序列
通過編寫自定義代碼工作流的選項擴展功能
13、API和開發(fā)人員
使用插件開發(fā)套件添加特殊功能或與其他系統(tǒng)集成
添加您喜歡的算法,數(shù)據(jù)庫或可視化
新功能
1、分析CRISPR編輯結果
輕松對齊,聚類和可視化CRISPR編輯實驗中的NGS讀數(shù)。通過您選擇的應用程序(包括HDR,NHEJ和基礎編輯器)分析變體的頻率及其蛋白效應。
2、增強型搜索選項
通過一個統(tǒng)一的界面快速訪問您的文檔,文件夾,分析工具和最近查看的項目。
3、密碼子優(yōu)化和反向翻譯改進
2020.2 –將鼠標懸停在優(yōu)化密碼子上,以獲取有關同義密碼子及其頻率的更多信息。
2020.1 –為模型生物定制密碼子優(yōu)化參數(shù)。
2020年–全新的密碼子優(yōu)化算法使您可以通過從蛋白質(zhì)或核苷酸序列開始生成新序列,來匹配所選生物體的密碼子用法。
4、分析CRISPR編輯結果的新工具
從CRISPR編輯實驗的結果中比對NGS測序并使其聚類
支持各種應用程序,包括HDR,NHEJ和基本編輯器
可視化與未突變參考序列相比的變體
分析變體的頻率及其蛋白效應
5、增強的搜索界面
使用文本查詢從單個統(tǒng)一界面實時搜索文件夾和文檔
搜索Geneious Prime中可用的分析工具(操作)的新功能
輕松訪問其他高級搜索選項
快速訪問最近查看的文件夾和文檔
6、密碼子優(yōu)化改進
有關優(yōu)化密碼子注釋的信息工具提示可在所選密碼子使用表中提供有關同義密碼子及其頻率的信息
現(xiàn)在,在通過選擇注釋優(yōu)化選擇的區(qū)域時,將考慮包括方向性和CDS codon_start信息在內(nèi)的注釋屬性
當以多個間隔優(yōu)化選定注釋時,跨間隔優(yōu)化選擇,并且允許密碼子跨越相鄰間隔
當優(yōu)化反向選擇區(qū)域時,反向互補序列現(xiàn)在已優(yōu)化
配置要求
Windows 7、8、8.1和10。請注意,Geneious Prime 2020及更高版本不支持32位Windows
注意:Windows不支持以下插件:TopHat,Velvet Optimiser
注意:使用Spades和Flye插件需要Windows 10以及用于Linux的Windows子系統(tǒng)
下載地址




































網(wǎng)友評論